Introducción a npphen
Autores: Matías Olea, José A. Lastra, Roberto O. Chávez (Diciembre 20, 2024)
Preambulo
El paquete de R "npphen" ha sido desarrollado principalmente para reconstruir la Fenología de la superficie (LSP; Land Surface Phenology en inglés) y para la detección de anomalías extremas utilizando series de tiempo satelitales. Sin embargo, las funciones incluidas en npphen permiten analizar todo tipo de series de tiempo numéricas. Ejemplos de series de tiempo raster que pueden ser analizadas provienen de distintos datos como GIMMS project, el U.S. MODIS Program, Landsat Program el programa de la E.S.A. Sentinel 2 o los datos de CR2MET que son productos climáticos chilenos. Ejemplos de series de tiempo numerias pueden ser el NPP (Net Primary Productivity) de una FluxNet Tower o el GI (Green Index) proveniente de una red de phenocams.
En este documento queremos facilitar el uso de este paquete de R demostrando en detalle lo que hacen de manera indivual cada una de las funciones y como deben ser aplicadas. Para esto, utilizaremos un demo de composites de "16-dias" del producto MODIS EVI (Enhanced Vegetation Index).
1 Preparando su sistema
Todos los paquetes de R necesarios (como terra y lubridate) estan automaticamente instalados junto npphen y son activados al llamar npphen:
| install.packages('npphen') library(npphen) |
| > ?npphen #para familiarizarse con las funciones |
2 Información satelital y procesamiento
Los datos son composites de "16-dias" del producto MODIS EVI de la ciudad chilena de Constitución, esta es una ciudad localizada en el clima mediterraneo de la zona central de Chile, lugar donde los bosques han sido afectados por un masivo incendio el año 2017. En total se utilizarán 491 imagenes obtenidas del datélite Aqua (producto MYD13Q1) el cual ha sido "depurado" utilizando sus bandas de calidad (QA bands).
Para este código ejemplo, utilizaremos las librerias "npphen", "terra" y "tidyverse".
2.1 Leer los datos EVI y guardarlos en un objeto SpatRaster
Primero leemos los datos EVI (multibanda) utilizando la función rast() del paquete terra. Luego, leemos el archivo CSV que contiene las fechas de cada banda.
|
evi <- rast('YourFolder/MYD13Q1_EVI_ts491_sample.tif') |
3. Fenología de la superficie (LSP) y detección de anomalías en series numéricas
Para poder comprender como funciona npphen y como lucen sus resultados, primero aplicaremos el algoritmo en una serie de tiempo EVI obtenida de un pixel. Podemos extraer la serie de la posición de un pixel en la matriz, o podemos extraer el valor con su coordenada utilizando la función extract(), y graficar los valores del índice para cada observación. Recomendamos repetir los siguientes pasos para un par de pixeles, especialmente si estamos trabajando en áreas heterogeneas. Para este código proveemos coordenadas, una para una plantación forestal y otra de un bosque deciduo.
|
xy_1 <- cbind(208567, 6077726) # Coord. de plantación forestal serie_1 <- terra::extract(evi, xy_1) %>% as.numeric() |
![]()
3.1 Trabajando con vectores numéricos (un único pixel): Phen
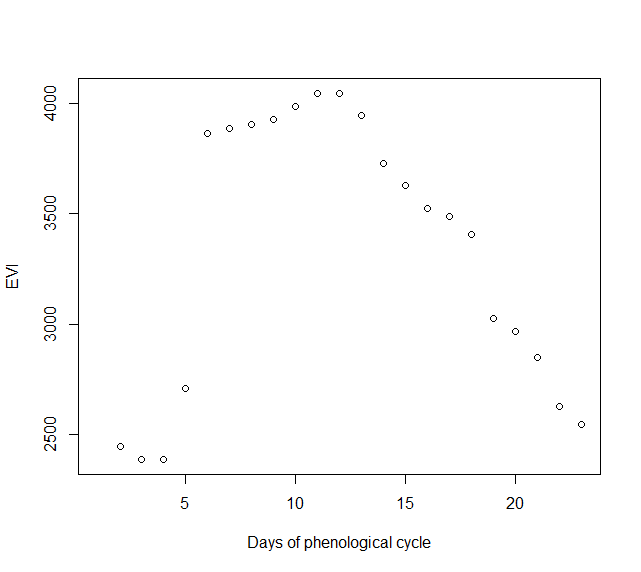
La función Phen() estima el ciclo fenológico de una serie de verdor. Requiere que tanto la serie como las fechas sean vectores. El argumento h indica el hemisferio geográfico y se usa para determinar el inicio del periodo de crecimiento (Growing Season). h=1 si el área de estudio está en el Hemisferio Norte (inicio de la temporada el 1ro de Enero), h=2 si está en el Hemisferio Sur (inicio de la temporada el 1ro de Julio). El argumento frequency define el número de muestras que se extraeran del total (365) de valores de la fenología, se debe seleccionar alguna de las siguientes opciones: 'daily' un vector con datos de los 365 días del ciclo, '8-days' un vector de 46 días del ciclo (i.e MOD13Q1 and MYD13Q1), 'monthly' un vector de 12 días del ciclo, 'bi-weekly' un vector de 24 días del ciclo (i.e. GIMMS) o '16-days' (por defecto) entregando un total de 23 (i.e MOD13Q1 or MYD13Q1). rge es un vector que continene el valor mínimo y máximo que corresponden al rango de la variable a estudiar. Sugerimos un rango teórico basado en los límites. Por ejemplo, en el caso de MODIS NDVI o EVI, los rangos van de 0 a 10.000, entonces rge = c(0,10000). El resultado de esta función es un vector numérico que contiene el valor esperado para el índice de vegetación para cada una de las fechas asociada al argumento frequency. Este valor esperado es la fenología de un periodo o temporada estandar de crecimiento (Growing Season), reconstruido a partir de toda la base de datos.
En este ejemplo utilizaremos la serie de tiempo del bosque deciduo.
|
PhenPix <- Phen(x = serie_2, # vector numérico con los valores del indice plot(PhenPix, xlab = 'Days of phenological cycle', ylab = 'EVI') |
3.2 Gráfico de la fenología y su RFD: PhenKplot
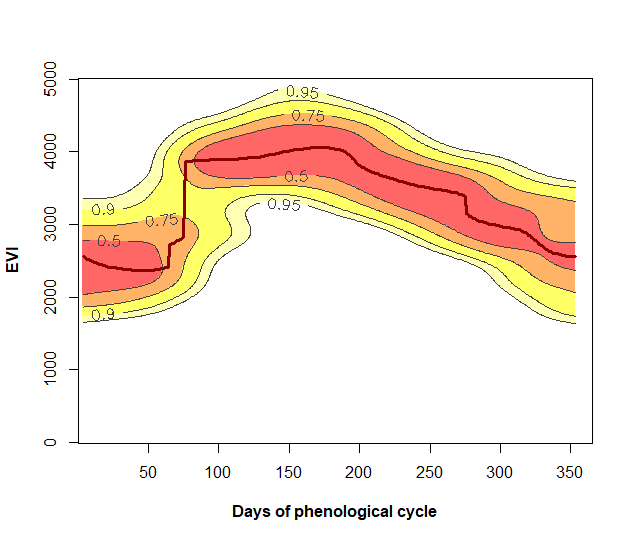
La función PhenKplot puede ser utilizada para visualizar su distribución de frecuencia de referencia (RFD; reference frequency distribution) de los valores de un índice en diferentes días del periodo de crecimiento. La linea roja es considerada la fenología de refencia, la cual es el resulado de la función Phen. Este plot no solo muestra la fenología para un pixel o serie en específico, sino tambien muestra con mayor detalle los rangos en los que se mueve el índice en el tiempo.
|
PhenKplot(x = serie_2, dates = table_dates$date,, h = 2, |
3.3 Detección de anomalias utilizando ExtremeAnom
Cuando nuestro objetivo no es recunstruir la fenología de la superficie sino detectar cambios en la vegetación asociados a un disturbio, podemos utilizar la función ExtremeAnom(). Esta función primero calcula el ciclo fenologico (como la función Phen) de un periodo dado para utilizarlo como referencia, y luego calcular anomalias en base a la diferencia entre el valor esperado (fenología de referencia) con un valor observado. Los argumentos refp y anop definen el periodo de referencia y el periodo al cual se le calcularán anomalias, respectivamente. El nuevo argumento output permite definir entre posibles resultados: 'both' (default) regresa un unico vector numérico con las anomalías seguido de sus valores de RFD. La extensión del resultado de 'both' es dos veces la extención del argumento anop, es decir, si definimos anop como c(1:10), la salida tendrá una extensión de 20L. La opción 'anomalies' solo entrega las anomalías, 'rfd' solo entrega los valores de RFD (que tan extremas son las anomalías) y 'clean' entrega solo los valores de anomalias extremas, es decir, anomalías que estén fuera de un rango de RFD (por ejemplo 0.90). Este umbral es definido por el usuario utilizando el argunento rfd . Este último, solo se aplica cuando output = 'clean'. Define los percentiles (de a 99) del rfd, con el cual las anomalías son catalogadas como extremas. Por ejemplo, si el rfd =0.90 solo las anomalías que caigan fuera del percentil 90 serán catalogadas como extremas, y el resto serán elimindas (se les asignará NA).
Para el siguiente ejemplo, utilizaremos la serie de tiempo de la plantación forestal con la marca del incendio del año 2017. Como periodo de referencia (refp) consideramos del año 2000 al 2010 (antes del periodo de Megasequía), y para el periodo de anomalías definimos de los años 2016 al 2018.
|
# Ejemplo de detección de anomalías con output='both' |
Para asegurarnos que escogimos bien los peridos, revisaremos en consola (esta parte es opcional)
| > # refp --------------------------------------- > table_dates$date[1] [1] "2002-07-04" > table_dates$date[196] [1] "2010-12-27" > # anop -------------------------------------- > table_dates$date[312] [1] "2016-01-09" > table_dates$date[380] [1] "2018-12-27" > # output length --------------------------- > length(c(312:380)) # anop [1] 69 > length(anom_plant) # output 'both' [1] 138 |
Ahora podemos graficar los resultados.
|
par(mfrow = c(2, 1), mar = c(2, 2, 1, 2)) |
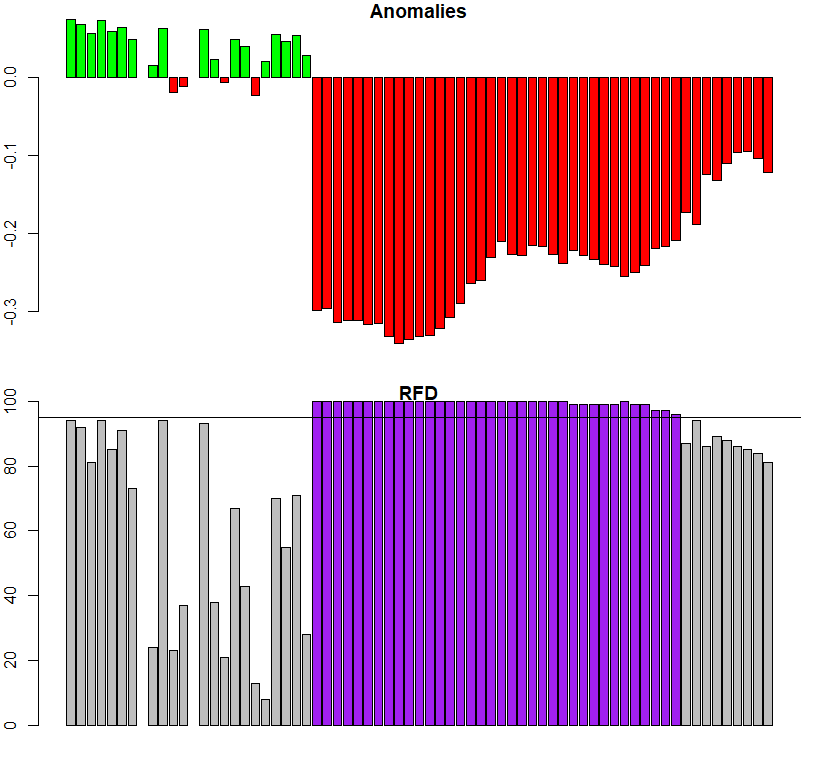
|
# Ejemplo de detección de anomalías con output='clean' |
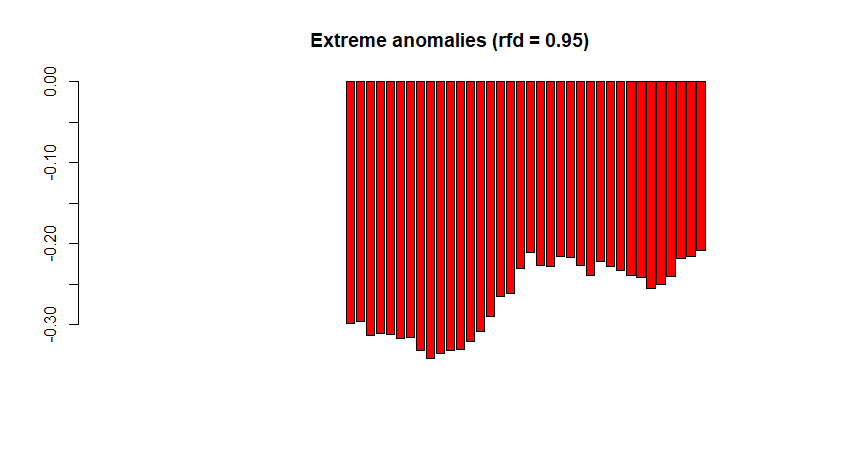
4. Trabajando con series de tiempo raster: PhenMap y PhenAnoMap
Ahora que hemos testeado las funciones para un único pixel, pasaremos a analizar la fenología de la superficie para toda nuestra área de estudio. Las funciones PhenMap() y ExtremeAnoMap() basicamente realizan la misma tarea que Phen() y ExtremeAnom() pero para todos los pixeles presentes en una serie de tiempo raster.
Adicionalmente, estas funciones incluyen el argumento nCluster que permite manipular el número de procesadores (cores) que queremos utilizar para el cálculo de cada función. Esto puede reducir significativamente el tiempo de computo, especialmente cuando estamos trabajando con grandes volumenes de datos.
Las salidas son raster multibanda con las mismas dimensiones espaciales y temporales que el raster de entrada. Es decir, si nuestro ejemplo posee 23 observaciones al año, la salida será un raster de 23 bandas.
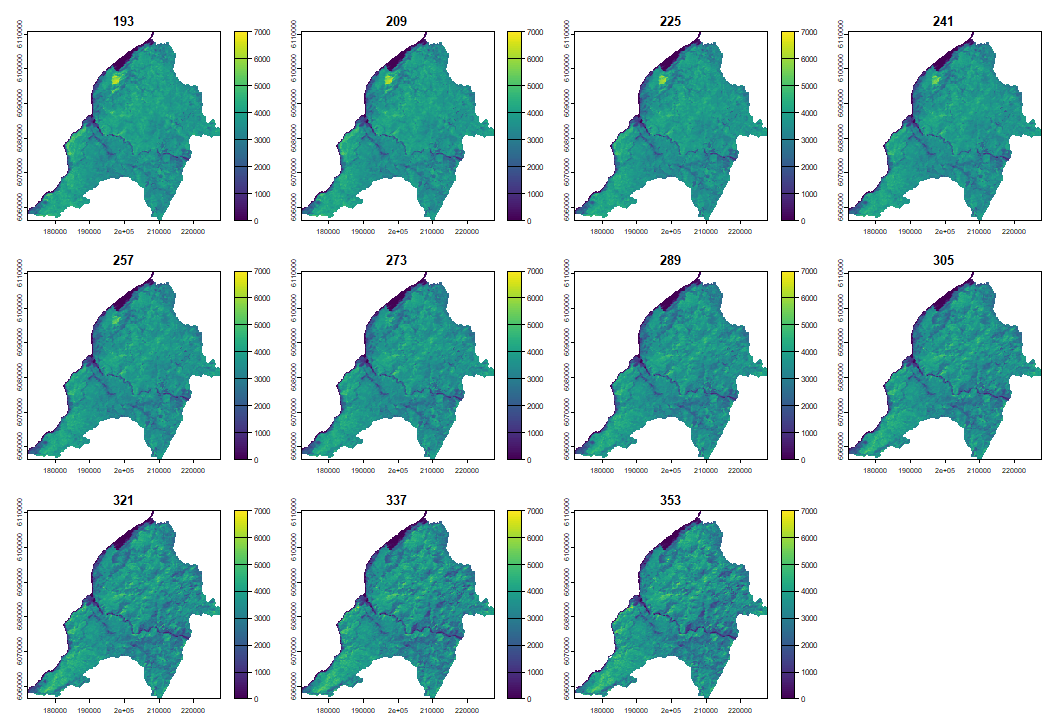
4.1 Reconstruyendo la Fenología de la Superficie con PhenMap
Cuando graficamos la fenología, podemos observar los patrones de enverdecimiento y empardecimiento asociados a la ganancia y perdidas de hojas verdes durante un año cuando corresponden a pixeles dominados por especies deciduas, cuando son especies siempreverdes los cambios no son tan evidentes.
|
PhenMap(s = evi, dates = table_dates$date, h = 2, |
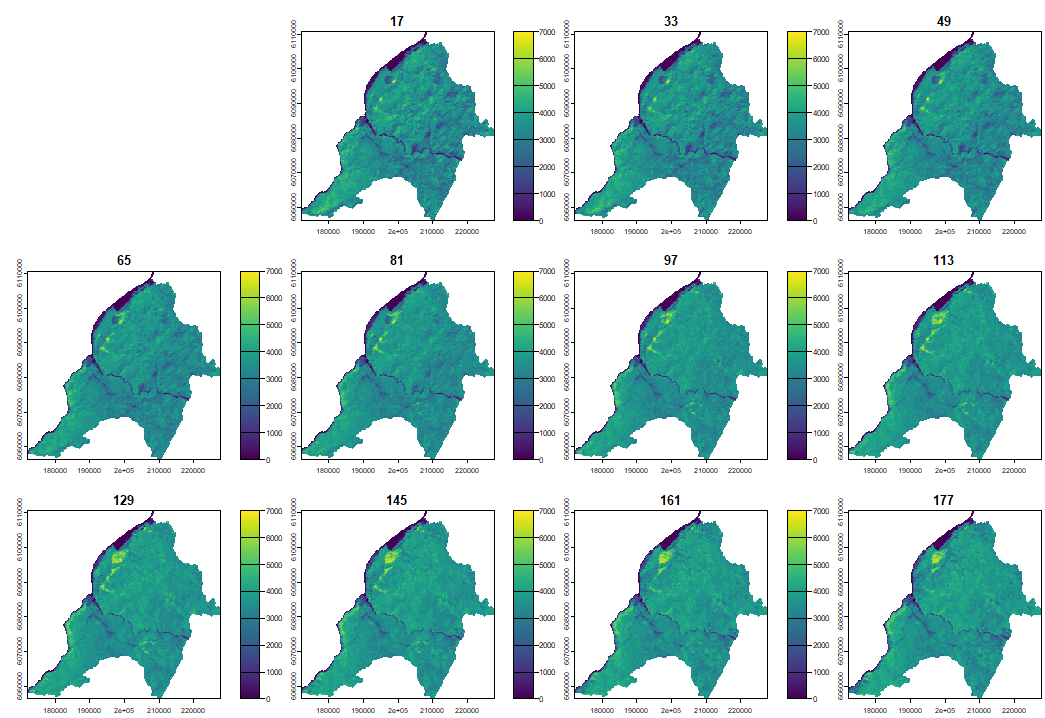
4.2 Detección de anomalías utilizado ExtremeAnoMap
ExtremeAnoMap() corresponde a ExtremeAnom() y puede ser aplicado a una serie de tiempo raster. Usaremos el mismo periodo de referencia que en los ejercicios anteriores (de la fecha #1 a la fecha #196), y un periodo de anomalias de 2016-2017 ("2016-07-03" al "2017-06-18"; de la fecha #323 a la fecha #345) para calcular las anomalías del incendio forestal del 2017.
|
ExtremeAnoMap(s = evi, dates = table_dates$date, h = 2, |
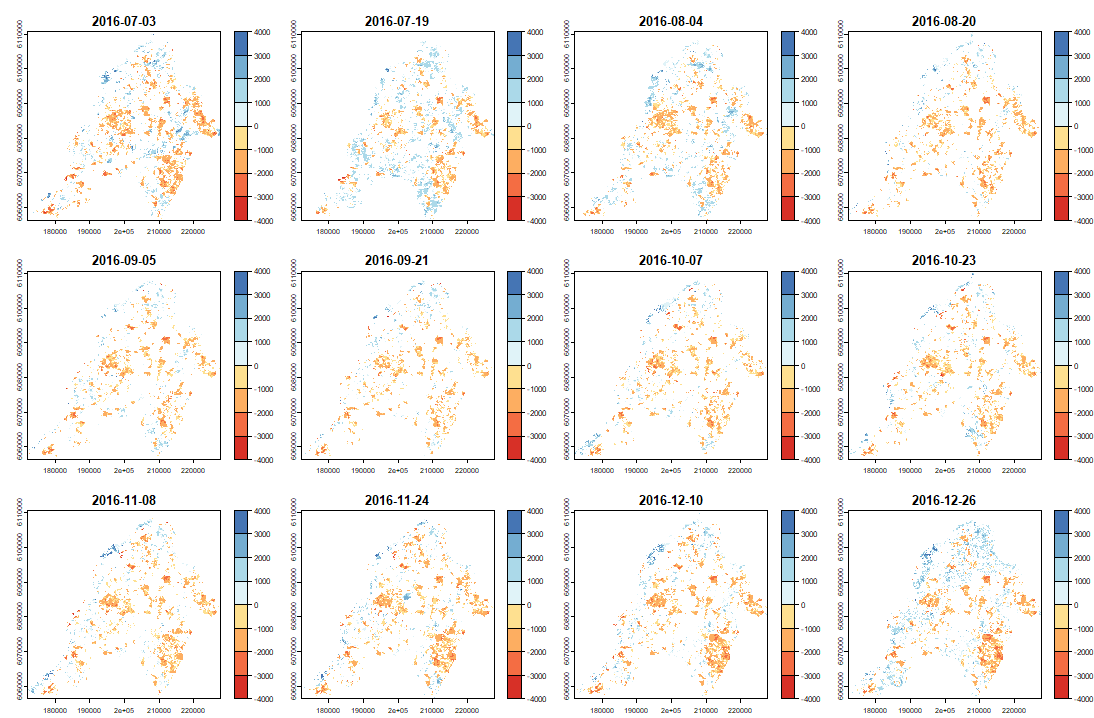
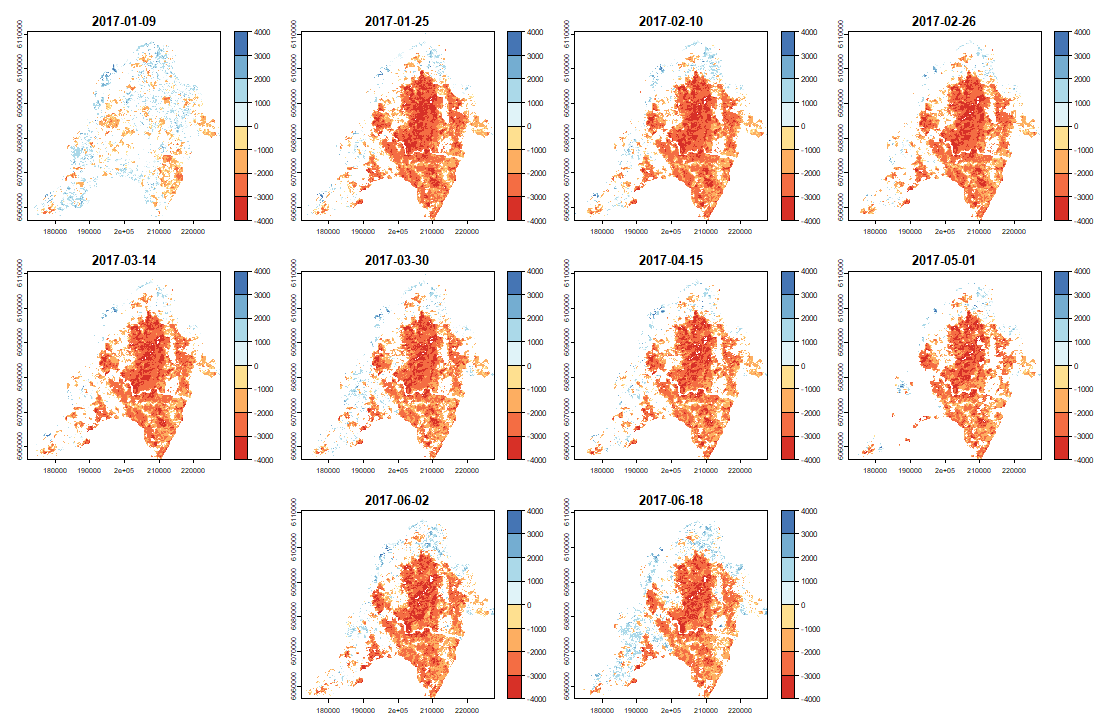
Ahora podemos mapear nuestras anomalías (primera mitad de las bandas)
|
anom.map <- rast('Anomalies_constitucion.tif') |
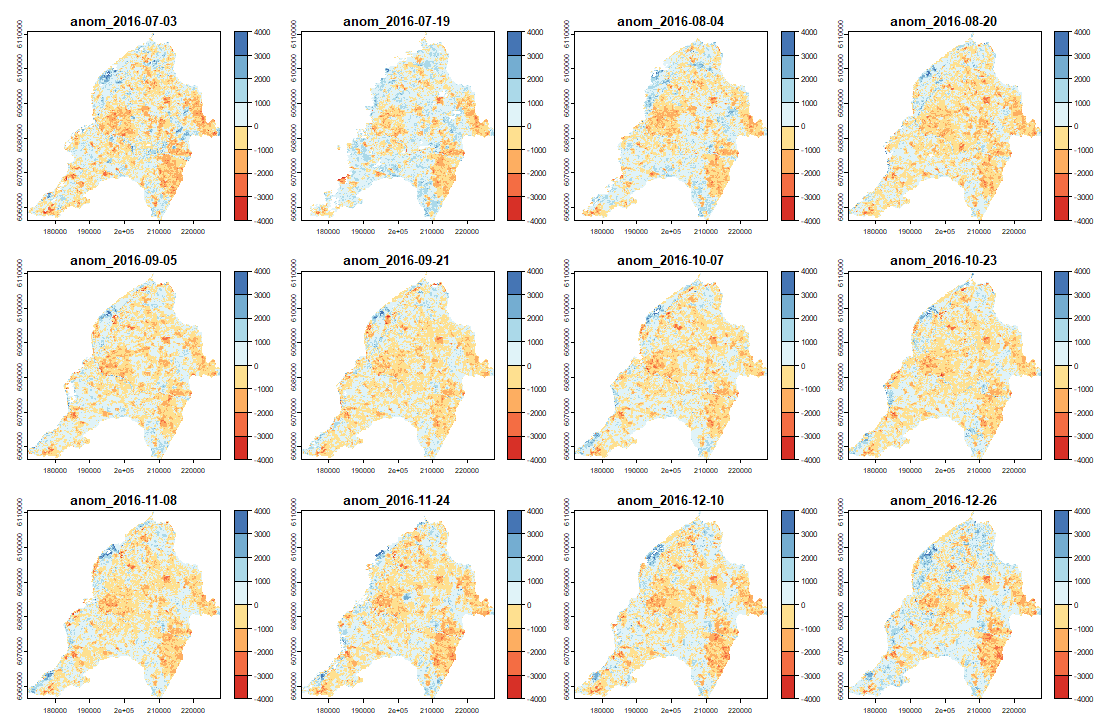

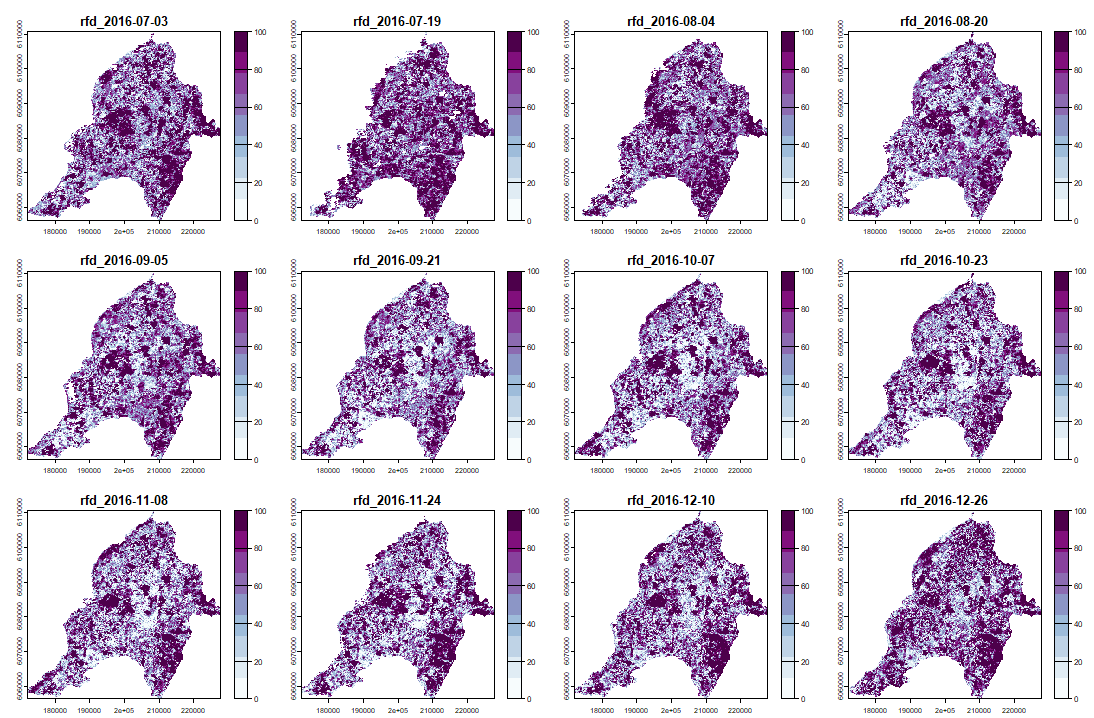
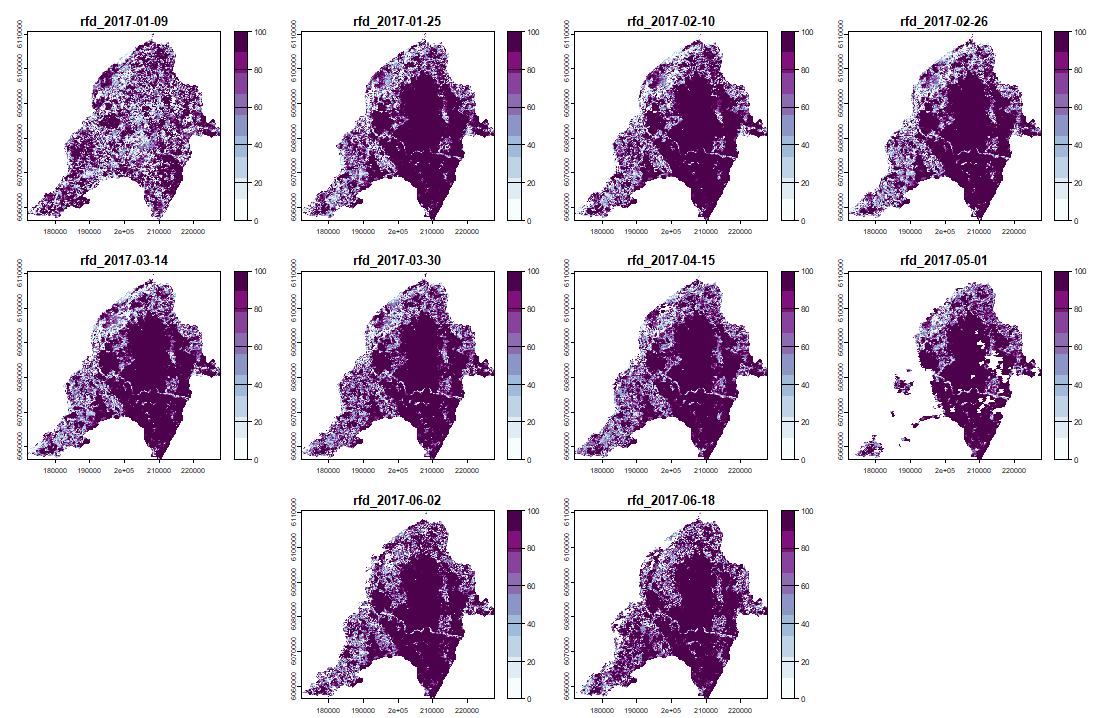
Y su RFD para cada fecha del "anop" (segunda mitad de las bandas)
|
anom.map <- rast('Anomalies_constitucion.tif') |
|
# Ejemplo utilizando el output = clean |
- Descargas
-
 Demo data
Demo data
-
 Demo table
Demo table